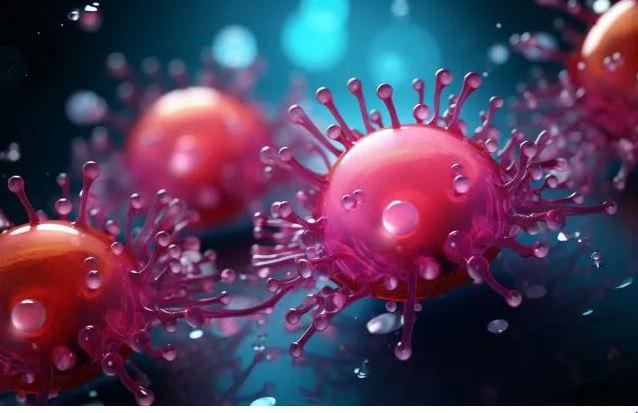
Se trata de los primeros 'virus vampiros' en estado salvaje que se identifican: usan esta técnica para poder replicarse en un huésped.
Un virus adherido a otro virus. Un equipo de científicos de Maryland ha observado este proceso en el que intervienen un virus "satélite" y un virus "colaborador". Estos virus auxiliares o colaboradores son virus que ayudan a otros virus en su reproducción y propagación. Además, proporcionan funciones primordiales, como crear capas protectoras o replicar material genético, de las que carecen los primeros. Si bien los investigadores saben desde hace décadas que algunos virus se aprovechan de otros virus, nunca se había observado un patógeno que se adhiere a otro exclusivamente para replicarse.
"Cuando lo vi, pensé: No puedo creerlo", dijo en un comunicado de prensa Tagide deCarvalho, primer autor de un estudio que anuncia el descubrimiento. "Nadie ha visto nunca un bacteriófago -o cualquier otro virus- adherido a otro virus".
Virus vampiro
El virus en cuestión es un bacteriófago, esto es, un virus que infecta bacterias, y también un virus satélite, que depende de otros virus "auxiliares" para completar su ciclo de vida. Para replicar su ADN, es preciso que los dos virus estén muy juntos. Este estudio ha demostrado que no solo están muy cerca, sino que uno se aferra al “cuello” de su virus auxiliar, como si de un pequeño vampiro se tratara para cumplir su proceso. Nadie se había dado cuenta de cuán cerca podían llegar algunos virus.
El trabajo estuvo dirigido por investigadores de la Universidad de Maryland del condado de Baltimore (UMBC), quienes descubrieron que algunas entidades parecidas a virus, conocidos como elementos genéticos móviles (MGE), se adhieren al cuello de sus ayudantes mientras infectan una población de células bacterianas. Al carecer de códigos para producir elementos críticos como una cápsula, estas secuencias dependen de la presencia de otros virus para fabricar su maquinaria. Tal y como podemos apreciar en la imagen, se trata del primer caso de 'virus vampiro' (en color morado) observado por científicos, que descubrieron que algunos agentes infecciosos se adhieren al cuello de otro (en color azul) para asegurar su ciclo de vida.
El equipo estudió una muestra de bacteriófago satélite (un virus que infecta las células bacterianas), incluida una especie de bacteria Streptomyces (la auxiliar) que se encuentra en el suelo. El satélite de la muestra es el primer caso conocido de un satélite sin gen de integración. Y no se trataba de un error. Repitieron el experimento múltiples veces. Los científicos observaron que el 80 por ciento de los virus auxiliares tenían virus satélite adjuntos, formando un matrimonio temporal. Los científicos incluso identificaron marcas que se asemejan a "marca de mordedura" en estos virus, lo que indica su estrecha vinculación. En 40 de los 50 experimentos fue así. Entre los 10 que estaban libres de satélites, todavía se podían ver restos deshilachados de fibras reveladoras envueltas alrededor de sus gargantas (muy parecidas a "marcas de mordeduras", dicen los investigadores).
"Este satélite ha estado sintonizando y optimizando su genoma para asociarlo con el ayudante durante, diría yo, al menos 100 millones de años", afirmó Ivan Erill, profesor de ciencias biológicas en la Universidad de Maryland.
Los investigadores descubrieron que el código genético utilizado por los virus satélite coincide con las instrucciones genéticas de los virus auxiliares. Esta coincidencia permitió que los virus satélite sobrevivieran dentro de sus huéspedes.
Los investigadores también pudieron analizar los genomas de los virus vampíricos, así como sus ayudantes y huéspedes, y descubrieron que la mayoría de los satélites tienen un gen que les permite integrarse en el material genético de la célula huésped.
El conocimiento adquirido a partir de estos estudios tiene el potencial de impactar en varios campos científicos y conducir a aplicaciones prácticas en biotecnología y medicina. El equipo espera que sus hallazgos inspiren trabajos futuros sobre este fenómeno inesperado para mejorar nuestra comprensión y tal vez explicar alguna extraña contaminación en la secuenciación de fagos.
Referencias:
deCarvalho, T., Mascolo, E., Caruso, S.M. et al. Simultaneous entry as an adaptation to virulence in a novel satellite-helper system infecting Streptomyces species. ISME J (2023). https://doi.org/10.1038/s41396-023-01548-0
Alam, K., Mazumder, A., Sikdar, S., Zhao, Y., Hao, J., Song, C., Wang, Y., Sarkar, R., Islam, S., Zhang, Y., & Li, A. (2022). Streptomyces: The biofactory of secondary metabolites. Frontiers in Microbiology, 13. https://doi.org/10.3389/fmicb.2022.968053.
Takasuka, T., Book, A., Lewin, G., Currie, C., & Fox, B. (2013). Aerobic deconstruction of cellulosic biomass by an insect-associated Streptomyces. Scientific Reports, 3. https://doi.org/10.1038/srep01030.
Rey, T., & Dumas, B. (2017). Plenty Is No Plague: Streptomyces Symbiosis with Crops.. Trends in plant science, 22 1, 30-37 . https://doi.org/10.1016/j.tplants.2016.10.008.